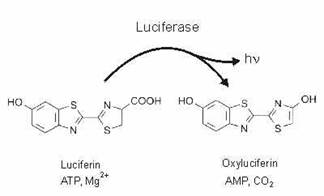

Gene reporter
Lo scopo del gene reporter è misurare il potenziale regolativo di una sequenza di DNA. Comuni geni reporter sono: β-galattosidasi, β-glucuronidasi e luciferasi. L'espressione si rivela mediante luminescenza, assorbanza o fluorescenza.
Gene reporter |
Metodo di rivelazione dell’espressione |
||
Luminescenza |
Fluorescenza |
Assorbanza |
|
Luciferasi |
+ |
+ |
|
β-galattosidasi |
+ |
+ |
+ |
β-glucuronidasi |
+ |
+ |
|
Secreted placental alkaline phosphatase (SEAP) |
+ |
|
+ |
Green Fluorescent Protein (GFP) |
|
+ |
|
Il metodo che utilizza il gene della luciferasi ha i vantaggi di essere molto sensibile, nessuna interferenza perchè la maggior parte delle cellule non ha attività di luciferasi, alta dinamica (possibilità di determinare bassi e alti livelli di espressione, la saturazione è a livelli molto alti di espressione), rapidità e bassi costi.
Il gene migliore è quello della lucciola nord americana Photinus pyralis, la proteina non richiede modificazioni post-traduzionali, non è tossica, funziona sia in cellule procariotiche che eucariotiche.


In generale il vettore contenente il gene reporter viene trasfettato nelle cellule. Dopo un certo tempo le cellule sono lisate, nell'estratto cellelare si introducono: luciferina (il substrato della luciferasi), Mg e un eccesso di ATP. L'enzima luciferasi eventualmente espresso, catalizza la carbossilazione ossidativa della luciferina causando luminescenza. Questo effetto può essere quantificato con un luminometro o un contatore a scintillazione. La quantità di luce rivelata correla direttamente con l'attività di binding del fattore di trascrizione.
Molti vettori disponibili in commercio hanno già copie multiple dei siti di binding per molti fattori di trascrizione noti.
Esempio di vettore
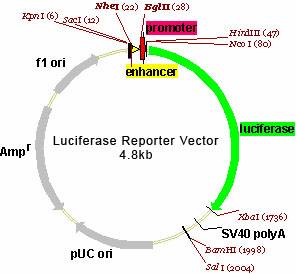
Ci sono copie multiple di un elemento enancher in cis, a monte di un promotore minimo TA e di una sequenza TATA box del promotore del gene ‘timidina chinasi’ del virus dell’Herpes simplex.
Queste sequenze si trovano tra i siti di restrizione NheI e BglII.
Il vettore contiene un gene per la resistenza all’ampicillina utile al clonaggio, un origine di replicazione, un origine (f1) per la produzione di DNA a singolo filamento.
PROTOCOLLO 1 |
PROTOCOLLO 2 |
 |
Il giorno prima della trasfezione si pongono le cellule (1-3 * 10^5) in 1 ml del loro normale mezzo di coltura contenente siero senza antibiotico. Fare varie aliquote in modo da avere dei duplicati. Il giorno successivo si dovrebbe avere una confluenza di circa 50% - 80%. |
Mettere 2 x 10^5 cellule per pozzetto (6-well) in 2 milliL di terreno di crescita completo con 10% FCS (siero fetale bovino). Incubare a 37°C con CO2 finchè circa il 70% delle cellule arrivi a confluenza. |
|
Per ciascun pozzeto contenete le cellule da trasfettare, aggiungere 0.5 microgr (1 microL) di vettore, aggiungere 50 microL di terreno di coltura senza siero. Ad ogni pozzetto aggiungere 3 microL di reagente di trasfezione, 50 microL di terreno di coltura senza siero, agitare e incubare per 5 minuti a temperatura ambiente, non incubare più a lungo. |
Preparare: |
 |
Dopo la trasfezione rimuovere il terreno di coltura, sostituire il vecchio terreno con il nuovo in cui c'è la sostanza che si deve comportare da stimolo per il fattore di trascrizione da testare. Per le cellule usate come controllo sostituire semplicemente il terreno con uno nuovo. |
Aggiungere 1 milliL di terreno di crescita completo con 20% FCS senza rimuovere la miscela di trasfezione. Aggiungere i composti (20 microL) che dovrebbero indurre l'espressione del fattore di interesse. |  |
A fine incubazione aspirare il terreno, aggiungere 50 microL a ciascun pozzetto (per 12-well) di buffer di lisi. |
Dopo 24 ore dalla trasfezione rimuovere la miscela di trasfezione e riaggiungere nuovo terreno con 10% FCS. Dopo 48 ore dall'inizio della trasfezione rimuovere il terreno e lavare 2 volte con 2 milliL di PBS. Eseguire la lettura con un luminometro. |
 |
| Riferimenti: TransLucent(TM) - www.panomics.com |
|